损伤和修复的模拟揭示了酶修复步骤对DNA存储的好处,特别是当数据以高存储密度(低物理冗余)和长时间存储在DNA中时。
近日,自然杂志发表了一篇由Linda C. Meiser,Andreas L. Gimpel,Tejas Deshpande,Gabriela Libort,Weida D. Chen撰写的文章名为“Information decay and enzymatic information recovery for DNA data storage”,翻译:用于DNA存储的信息衰减和酶信息恢复
论文摘要:“合成DNA因其较高的理论存储密度和预期的长存储期限而被提议作为数字信息的存储介质。然而,在所有环境储存条件下,DNA都会经历缓慢的化学衰变过程,导致DNA链被切口(断裂),并且存储在这些链中的信息不再可读。在这项工作中,我们设计了一种酶修复程序,该程序适用于读出前的DNA库,并且可以部分逆转损伤。通过对腐烂过程的化学理解,受损部位 3' 端的悬垂被确定为阻碍修复通过基础切除修复 (BER) 机制。可以通过核酸/无嘧啶核酸内切酶 I (APE1) 去除梗阻,从而能够通过 Bst 聚合酶和 Taq 连接酶修复水解损伤的 DNA。损伤和修复的模拟揭示了酶修复步骤对DNA数据存储的好处,特别是当数据以高存储密度(=低物理冗余)和长时间存储在DNA中时。
内容:
存在于所有生物体中的基因组DNA可能会受到外在和内在因素的破坏,包括暴露在阳光下,氧化或水解以及DNA链中的病变等因素.尽管有自然的校对机制,但其中一些病变和错误可能仍然无人看管,如果不修复,可能会诱变。
已知三种基本的细胞防御机制,即直接修复、碱基切除修复(BER)和核苷酸切除修复(NER),其中特定酶修复自发DNA病变。这些病变可能是由阳光、氧化、水解或暴露于小分子引起的。在没有大量外部DNA损伤剂的情况下,大多数DNA病变通过碱基切除修复途径修复,该途径已由Dianov和Lindahl于1994年重建。使用纯化的酶,林达尔因此共同获得了2015年的诺贝尔化学奖.
由于鉴定了碱基-切除-修复途径中涉及的酶,修复酶恢复降解DNA的质量不仅用于理解基因组DNA修复,而且用于标准的分子生物学常规,并且还被提议用于改进古代DNA的分析,法医样本的基因分型以及食物的追踪,仅举几例.然而,酶修复的一种尚未受到任何关注的应用是在DNA数据存储应用中修复合成DNA。
对于DNA数据存储,数字文件(比特)被翻译成核苷酸(nt),然后可以合成并存储数千年由于合成和读取非常长的DNA链的限制,数据通常存储在许多相对较短(约150 nt)寡核苷酸的池中.除了对实际数据进行编码外,每个寡核苷酸还包括一个用于信息组织的索引和扩增引物,可实现随机访问以及用于读出的寡核苷酸的处理(测序制备)。该技术通常使用纠错码,计算冗余信息并将其附加到原始数据中,以便可以补偿读/写和存储错误 .因此,这种纠错码可以纠正序列(内部代码)中的单个基本错误(例如突变),以及完整序列(外部代码)的丢失。虽然这种纠错码能够完美地恢复存储的数据,但它们的代价是必须写入(=合成)比没有错误时所需的核苷酸和寡核苷酸更多。这并不意味着必须以更大的规模合成DNA库(即每个寡核苷酸有更多的拷贝),而是必须合成更多独特的寡核苷酸以实现纠错(例如,更大的外部代码冗余)。其成本与预期错误数直接成比例.DNA合成是目前更广泛采用DNA数据存储的瓶颈,因此应尽量减少合成工作。我们在最小化错误以及必要的数据冗余方面的特定兴趣在于实际的DNA存储过程本身。
在这项工作中,我们分析了合成DNA的衰变过程,预计该过程将在分子水平上的DNA数据存储场景中发生。我们将DNA切口确定为导致最大数据丢失的过程,并提出酶修复以寻找扭转这种数据丢失的潜在解决方案。该解决方案提出了数据存储应用中合成DNA的信息恢复过程。
在储存过程中,DNA可能会受到多种压力,包括紫外线照射,氧化,水解,烷基化,电离辐射或机械剪切,但与实际储存形式无关,水解已被确定为主要的衰变反应.这是由于大气湿度几乎无处不在,以及DNA与水而不是氧气的反应速率相对较高。此外,水是所有DNA处理步骤的标准培养基,因为DNA不溶于水以外的任何其他溶剂。
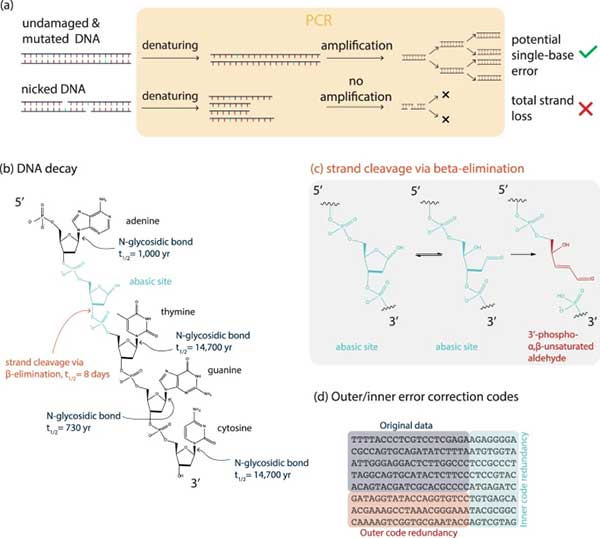
在DNA衰变方面,有两种衰变模式特别令人感兴趣:单个碱基的突变和DNA骨架的切口(=链断裂)的形成。虽然过去已经描述了突变的性质,从信息读出的角度来看,DNA切口尤其成问题,因为切口的DNA不能通过聚合酶链反应(PCR)扩增,这是在随机访问和DNA读出(测序制备)例程中通常采用的程序(图)。任何包含至少一个单链断裂的DNA链都不会被扩增,因此不会被读取。在信息完整性的背景下,突变可能导致多达 2 位信息的丢失,而缺口可能导致超过 100 位数据丢失(假设序列长度超过 50,通常是这样)。虽然这些损失可以通过纠错码来减轻,但突变与缺口的成本不平衡仍然存在: 纠错码校正突变碱基的成本相对较低:一个替换错误至少需要两个冗余符号(内部代码,图)),一个擦除(缺失核苷酸)至少需要一个冗余符号,一个符号的大小可以不同,常见的选择是每个符号 3 nt).然而,整个序列的丢失(由于单个DNA缺口)的成本要高得多,因为序列的整个信息都会丢失。从纠错的角度来看,这是昂贵的,因为每个缺口的校正至少需要一个完整的冗余DNA链(外部代码冗余,图)。). 具体来说,切口的预期成本比突变的成本高出序列长度的一半左右。因此,在实践中,缺口的成本通常比突变的成本高25-100倍。因此,我们对研究储存过程中DNA的切口感兴趣。
DNA数据存储过程中的衰变途径图示
研究结果
因此,我们通过观察两组不同的DNA来研究水解发生的DNA损伤,一组代表单个寡核苷酸(模型DNA),另一组包含7373个寡核苷酸,共同编码115,394字节的数据(补充表1)。我们将DNA在25°C和30°C的水中自然老化长达40天,并通过qPCR定量可扩增DNA的量。虽然由于DNA对水的部分保护,各种DNA保存方法可以使较慢的水解,但可以预期主要的衰变促进步骤仍将包含相同的水解反应,但速率明显低于水溶液。
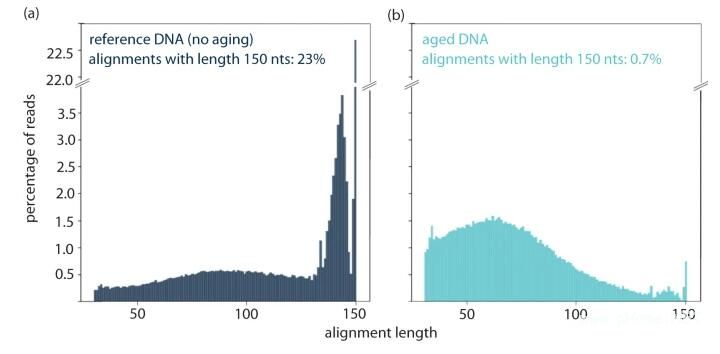
为了研究切口过程,我们使用Illumina的iSeq100通过ssDNA测序测量了衰老DNA的片段化。为此,我们使用了略微不寻常的商业样品制备程序(Swift Accel-NGS 1 S Plus),该程序可以分析切口ssDNA片段。与DNA数据存储中应用的传统扩增子测序相比,该制备程序依赖于专有的自适应步骤,并且不需要PCR或连接作为第一个样品制备步骤。因此,此过程可以读取不完整长度的序列(=片段)。为了进行分析,根据片段大小对单链DNA序列进行比对和比较。这确保了合成或测序错误不会影响片段分析,并且可以通过查看片段大小分布直接评估切口过程。图 2 显示了编码 115 kB Jpg 图像的文件的片段大小分布,由未损坏文件的 7,373 条独特 DNA 链组成(图2a)和老化的文件(图2a)。). 每个设计,每个序列的全长为 150 nt。我们观察到,在未受损的DNA文件中,近23%的比对具有正确的链长度,而在样品老化后,只有0.7%的比对具有正确的链长度。片段大小分布如预期的那样变化:老化的文件包含许多片段长度为 50-70 nt 之间的小片段,并且没有特定片段长度的普遍存在。这一发现支持了一般假设,即在控制良好的条件下,DNA衰变是一个随机过程。
在这项工作中,我们分析了用于DNA数据存储应用的合成DNA的衰变和修复过程。我们提出了作为DNA衰变的一种形式的切口过程中化学过程的分子分析,并重点关注由于水解引起的切口。实验条件代表了DNA处理步骤中的真实场景,并说明了水的威胁(以处理介质的形式,以及储存过程中的湿度形式)。我们发现,当链被切开时,水解会导致3'末端的分子阻塞,从而导致PCR变性步骤中的链损失。由于链损失只能通过额外链的冗余来补偿,因此与单核苷酸突变相比,切口是一种非常昂贵的损伤形式。因此,我们设计了一种含有三种单酶(APE1,Bst聚合酶和Taq连接酶)的酶溶液,旨在去除切口后的阻塞性醛基团,随后密封切口以修复DNA链,以便再次进行PCR扩增。我们发现修复可以恢复信息,在低损伤状态下恢复率更高,恢复率超过30%。将我们自行设计的修复组合与商业试剂盒(即PreCR®修复组合)进行比较,我们的修复可以更便宜,更快,更有效地回收DNA。
在低物理覆盖率下,丢失DNA链会导致数据(包含在该链中)丢失并降低读数。因此,我们模拟了物理和逻辑冗余的要求,以预测和控制DNA数据存储的编码过程,以便仍然可以读出。这在转向高密度存储时特别有用,DNA特别适合高密度存储。
将来,当将传统的存档过程转换为包括DNA作为存储介质时,DNA的酶修复可能对数据档案特别感兴趣。当存储时间未知时,信息恢复方法是有益的,如果标准解码和读出失败,则提供恢复选项。
英文原文地址:https://www.nature.com/articles/s42003-022-04062-9
声明: 此文观点不代表本站立场;转载须要保留原文链接;版权疑问请联系我们。